© Stevenn VOLANT / Amine GHOZLANE / Javier PIZZARO CERDA / Juan Jose QUEREDA TORRES / IPL / CNRS Images
Référence
20170074_0012
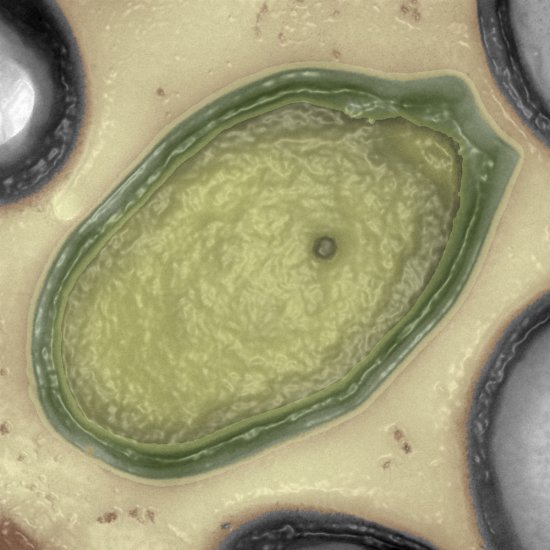
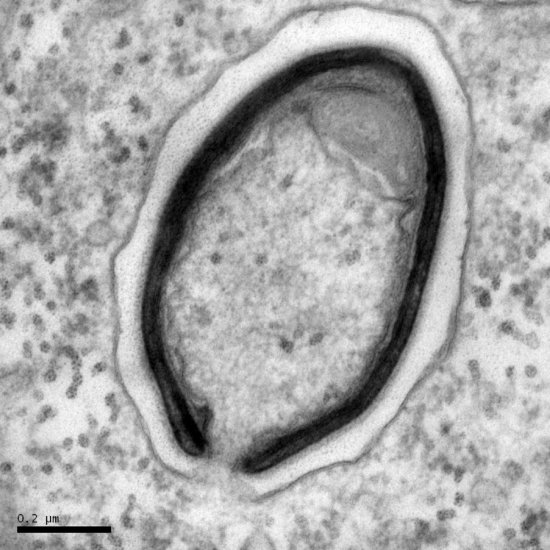
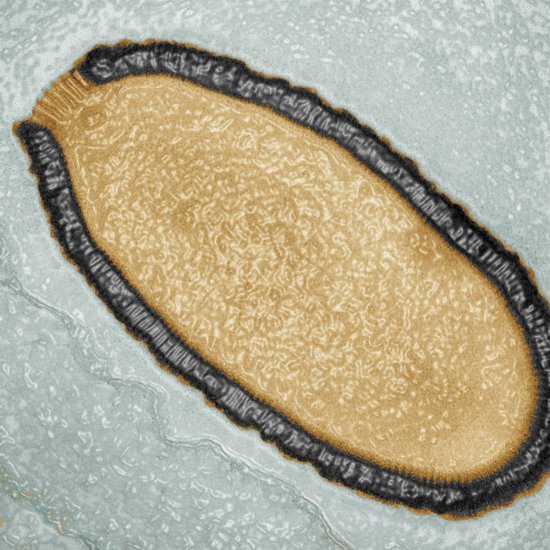
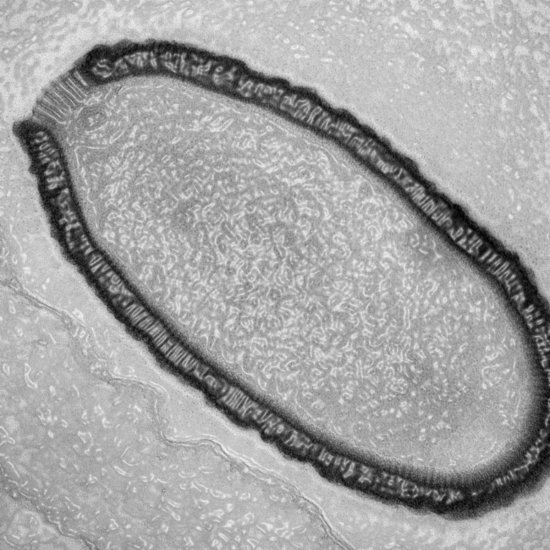
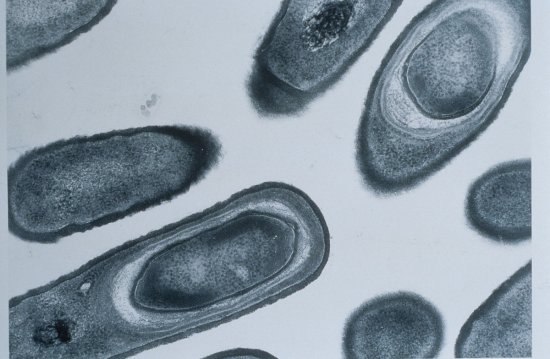
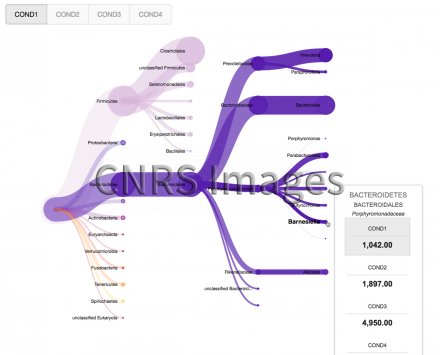
Arbre d’abondance métagénomique d'un microbiome intestinal de souris
Arbre taxonomique d’abondance dynamique obtenu par analyse de métagénomique quantitative. Cette analyse permet d’identifier des associations entre un microbiome, différentes conditions environnementales et des individus présentant des caractéristiques particulières (maladies, origine géographique…). Ici, les chercheurs ont étudié le microbiome intestinal de souris infectées par la bactérie "Listeria monocytogenes". Dans cette expérience l’abondance des populations de bactéries "Alloprevotella" et "Allobaculum" est corrélée àl’expression du gene isoleucine S (LLS). Cette visualisation a été générée par le biais du logiciel SHAMAN (SHiny Application for Metagenomic Analysis). Les récents développements des nouvelles technologies de séquençage permettent d’étudier ces organismes à l’échelle du génome. Pour "Listeria" cela permet par exemple l’identification de nouveaux facteurs de virulence bactériens notamment lors de la phase intestinale de l’infection.
L’utilisation des médias visibles sur la Plateforme CNRS Images peut être accordée sur demande. Toute reproduction ou représentation est interdite sans l'autorisation préalable de CNRS Images (sauf pour les ressources sous licence Creative Commons).
Aucune modification d'une image ne peut être effectuée sans l'accord préalable de CNRS Images.
Aucune utilisation à des fins publicitaires ou diffusion à un tiers d'une image ne peut être effectuée sans l'accord préalable de CNRS Images.
Pour plus de précisions consulter Nos conditions générales