© Violette DA CUNHA / Philippe GLASER / Pierre LECHAT / Erika SOUCHE / Ivan MOSZER / IPL / C3BI / CNRS Images
Référence
20170074_0013
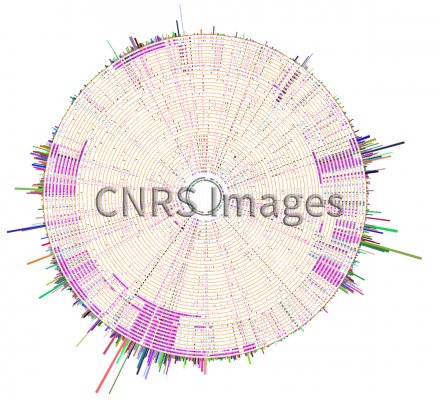
Représentation circulaire de la distribution et de la recombinaison de SNPs
Représentation circulaire de la distribution et de la recombinaison de SNPs (qui sont des variations mineures du génome au sein d’une population ou d’une espèce, à l’origine de différences morphologiques mais aussi de certaines prédispositions à des maladies). Ici, les chercheurs ont comparé des lignées de clonotype cc1 du groupe B de "Streptococcus agalactiae" (GBS) avec celle de la bactérie "Streptococcus agalactiae", principale cause d'infections néonatales bactériennes. Les récents développements des nouvelles technologies de séquençage permettent d’étudier ces organismes à l’échelle du génome. Dans le cas de "Streptococcus" cette étude a permis de reconstruire l’histoire évolutive récente des clones majeurs responsable des infections. Les mutations intergéniques sont présentées en noir, les mutations synonymes sont en rose et les non synonymes sont en bleu. Les régions de recombinaison sont les zones les plus denses en SNPs. Cette visualisation a été générée avec le logiciel SynTView (Lechat P, Souche E, Moszer I).
L’utilisation des médias visibles sur la Plateforme CNRS Images peut être accordée sur demande. Toute reproduction ou représentation est interdite sans l'autorisation préalable de CNRS Images (sauf pour les ressources sous licence Creative Commons).
Aucune modification d'une image ne peut être effectuée sans l'accord préalable de CNRS Images.
Aucune utilisation à des fins publicitaires ou diffusion à un tiers d'une image ne peut être effectuée sans l'accord préalable de CNRS Images.
Pour plus de précisions consulter Nos conditions générales